强悍的结构预测软件AlphaFold2已经开源,程序代码部分基本可以随意使用包括分发、修改等,深度学习的模型不得用于商业化用途,对学术界来说是个重磅利好。
AlphaFold2开源地址:https://github.com/deepmind/alphafold
AlphaFold2预测的结构数据库:https://www.alphafold.ebi.ac.uk/
安装过程大致如上面那个网址描述的一般。网上也有更详细的教程:
-
通过Docker安装和运行的:AlphaFold (ver.2) installation --Qiita ,看不懂日文的没关系,翻译成英文就好了。
-
通过Conda安装非Docker版的,如:https://github.com/kalininalab/alphafold_non_docker 和 https://github.com/kuixu/alphafold
两种版本我都试过,都可行,个人更喜欢第二种方法,因为改代码更加方便。
有一点需要特别注意: 一定要根据自己的显卡型号选择兼容的驱动、CUDA和CUDNN,同时CUDNN也要兼容TensorFlow 2.5。强烈建议根据官网教程进行安装驱动和CUDA:https://docs.nvidia.com/cuda/cuda-installation-guide-linux/index.html
现在也有在线版的AlphaFold2可供使用:
-
https://colab.research.google.com/github/sokrypton/ColabFold/blob/main/AlphaFold2.ipynb
-
https://colab.research.google.com/github/sokrypton/ColabFold/blob/main/AlphaFold2_complexes.ipynb#scrollTo=JPWfhGssZdTb (复合物结构预测,缺点是不能使用Amber Relax)
-
https://colab.research.google.com/github/deepmind/alphafold/blob/main/notebooks/AlphaFold.ipynb
以上都是通过MMseqs2生成多序列比对结果(MSA),而AlphaFold2社区版(github上公布的那个版本)生成MSA使用的是HHblits/HMMer算法。由于预测的精度极大的取决于找到的同源序列数量的多寡(越多则共进化信息越丰富),因此上面服务器跑出来的结果和AlphaFold2社区版在精度上可能会有差异。如果有条件的话,建议还是使用社区版进行预测。
说到条件,我们就简单的说说搭建本地服务器需要哪些硬件和软件条件:
-
尽量使用Linux系统,比如ubuntu 18或者ubuntu20等。win10总是会因为各种权限问题导致各种莫名其妙的错误(没有真正测试过,通过安装过其他大型软件得出的经验)。
-
尽量使用固态硬盘SSD,而且是使用NVME协议的,容量3TB~4TB。市面上比较便宜的是2TB的,4TB的比较少,而且很贵。因此如果要买2块2TB的,需要看看主板是否有足够多的M.2接口。尽量不要用机械硬盘,尽管它们很便宜4TB才600左右,但是速度确实够慢。
-
CPU尽量12核心以上
-
内存尽量48G以上
-
支持CUDA11的Nvidia显卡。这个丰俭由人,有钱的可以上Tesla系列,钱包捉急的GTX1660也可以。显卡在整个建模过程中的最后一步用上,占比整个运行时间比较短,所以如果耐心还可以,任务也不是特别多的可以上个便宜的显卡。
使用体验
了解AlphaFold2大致原理的可能都知道,AlphaFold2的预测精度很大程度上依赖于共进化信息的丰富程度。所以如果数据库中同源序列越多,预测的精度一般越准确。同源结构的存在与否、同源结构与目标序列的同源度的高低影响不是那么大。有几类蛋白AlphaFold2预测的还是不够准的:
-
无序蛋白(Intrinsic Disordered Protein);所谓无序,就是结构构象不稳定,存在多种构象的可能。
-
有时候膜蛋白预测的也不行。
-
同源序列极少的蛋白,比如设计的一个全新的蛋白。
-
抗原抗体复合物结构,这个就不存在什么共进化信息。
下面我们举几个例子来看看AlphaFold2的效果
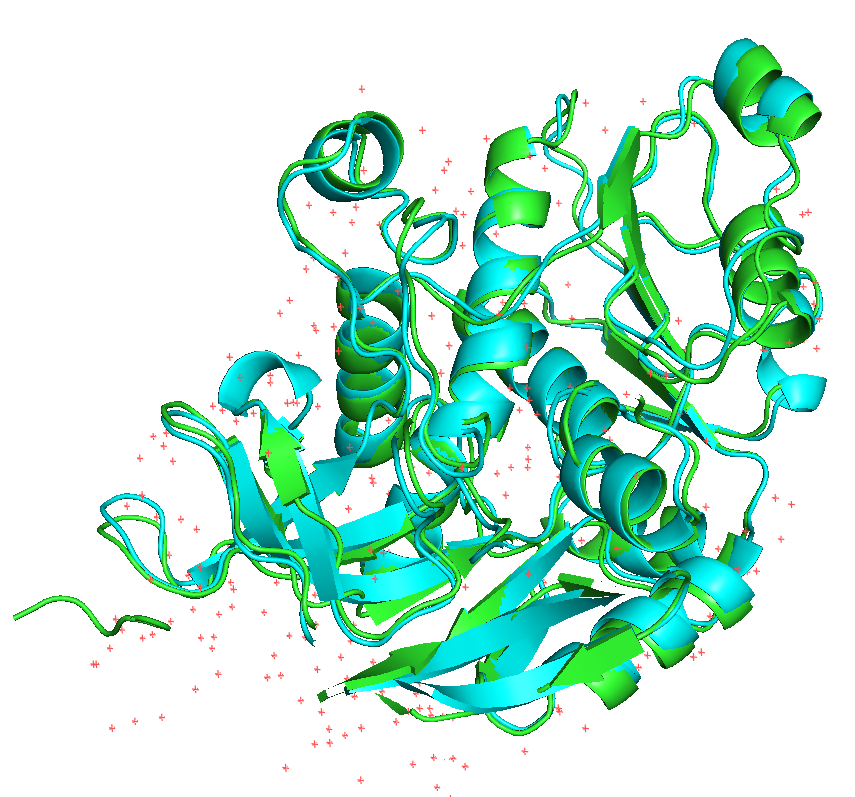
图1. 这张图显示的是一个327aa的蛋白与同源结构最高identities=30%的结构预测结果(青色为预测的,绿色的为我们解析出来的结构)。两个结构RMSD=0.86埃,预测结果非常好。
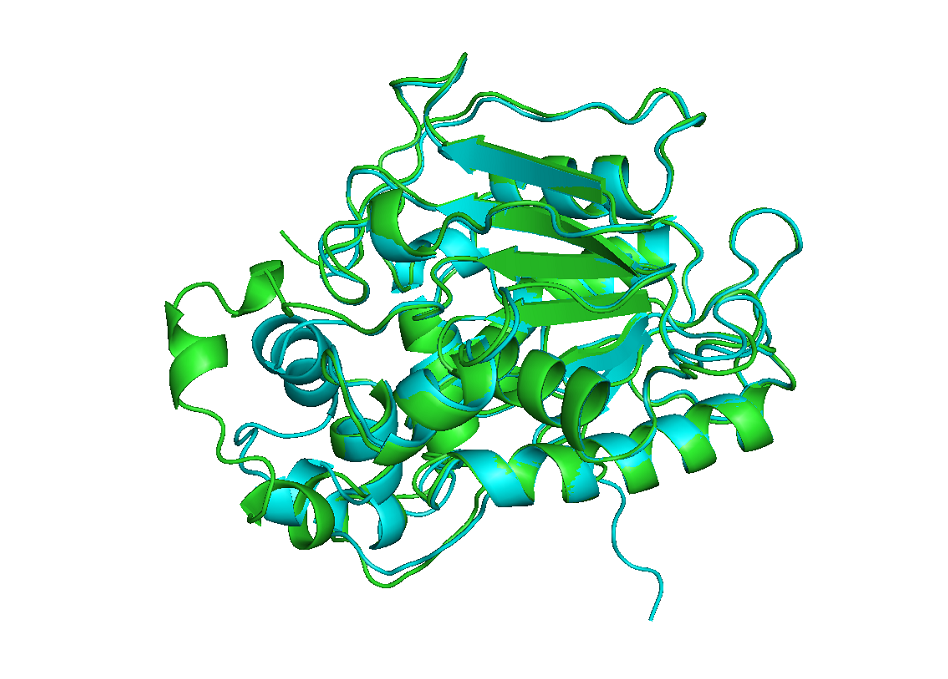
图2. 这个预测的蛋白长度297aa,最高identities 35%;两个结构的RMSD为0.56埃(青色为预测的,绿色的为我们解析出来的结构)。结果依然非常棒!
评价结构预测可信度有一个指标,叫pLDDT,由AlphaFold2给出。pLDDT的指示意义如下:
Very high (pLDDT > 90)
Confident (90 > pLDDT > 70)
Low (70 > pLDDT > 50)
Very low (pLDDT < 50)
上面两个结构的pLDDT都非常高,都在90以上。下面的这个结构pLDDT只有60左右:
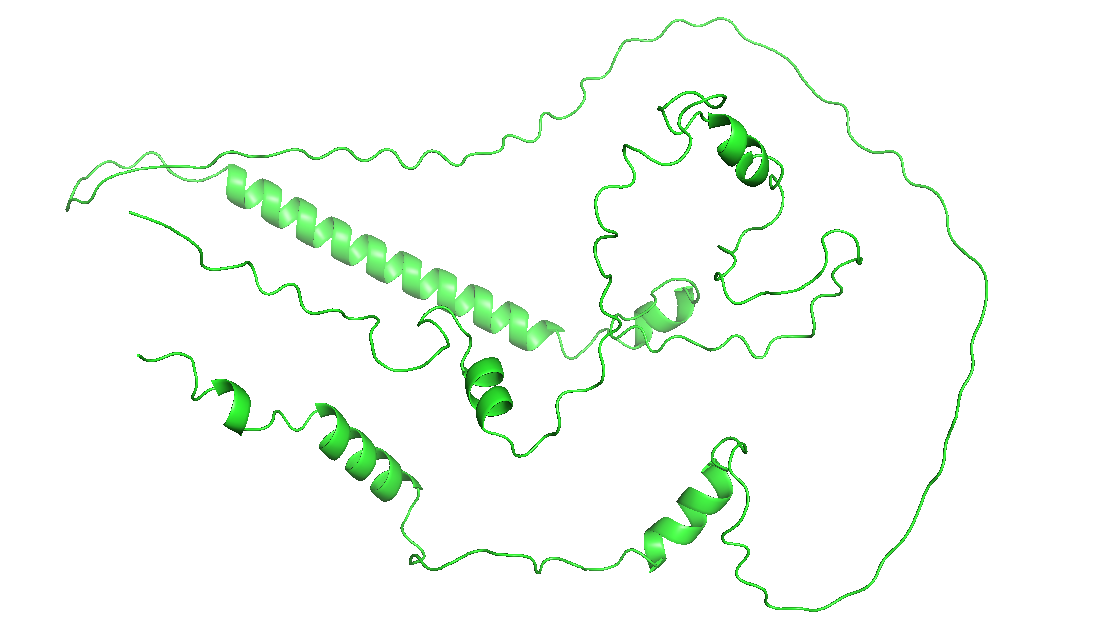
图3. 这个蛋白找到的同源序列非常少,pLDDT只有60,结果不可信。
是不是pLDDT高的就一定准确无疑呢?答案是否定的。请看下面的案例:

图4. 蛋白长度440aa,与同源结构最高identities为36%;pLDDT=95.8. 光看pLDDT,觉得这个结构铁定非常准确了。但是这个结构与真实结构相差非常大,请看它的真实结构:
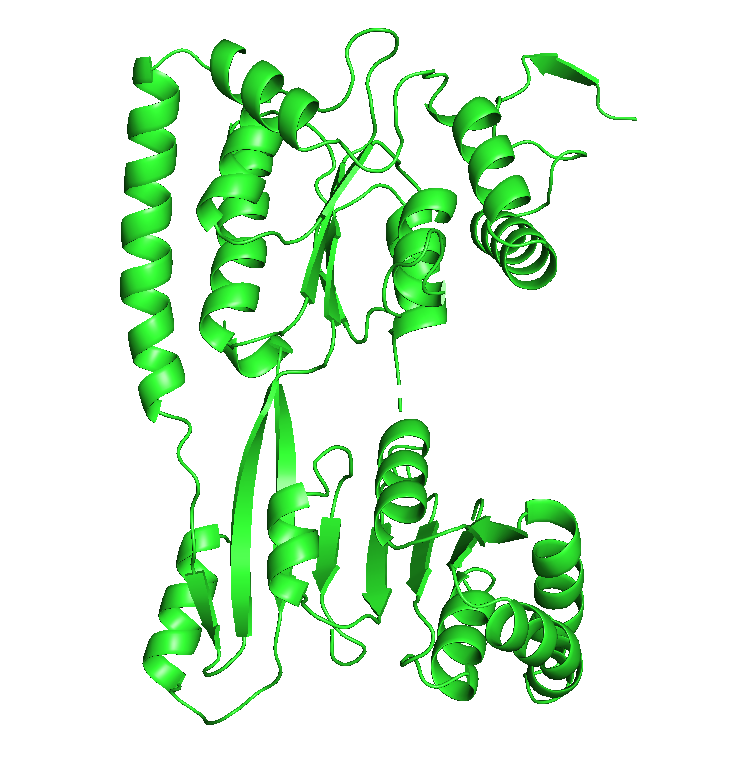
图6. 我们解析出来的晶体结构。从整体结构上来看,尽管pLDDT非常高,但是结构却完全是错的。不过尽管如此,预测的结构中有一部分是非常准确的,可以用来做分子置换求解相位。因此有了如此神器,导师再也不用担心蛋白晶体结构的相位求解和冷冻电镜的建模问题了。
上面做完了单体蛋白的预测,我们也尝试了复合物结构的预测。无论是同源多聚体还是异源多聚体,理论上来说共进化信息依然是可以在数据库中找到的,只要存在数量足够的同源序列。
我们首先测试了抗体Fab复合物结构的预测:
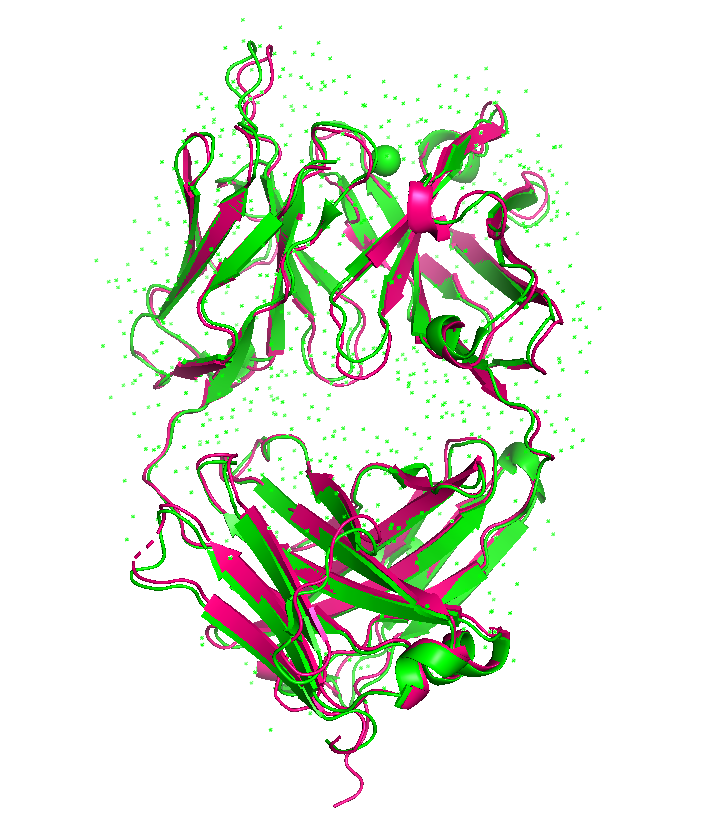
图7. 绿色的为3PP4结构,红色的为预测结构,RMSD=0.86埃。可以从图中很明显的发现抗体的CDR3预测的还是非常棒的。
未完待续。。。
2021-08-02更新:最近几天用alphafold测试了好多种蛋白结构建模,懒得贴结果了。直接说结论:1. 对于有着丰富共进化信息的蛋白质,预测结果通常会比较精准;2. pLDDT通常能衡量建模的质量,但是总有例外,高pLDDT依然有可能是错误的模型;3. 蛋白loop区的预测非常有挑战,特别是长loop区,不过这些区域即使通过实验手段也很难确定它的结构(比如晶体学往往看不到电子密度);4. 对复合物的预测有时候和真实结构差异非常大,但有时又会带来惊喜,这里说的复合物包括同源多聚体和异源多聚体;5. 比较难用于定点突变的结构模型预测,试想有些突变是会让蛋白去稳定的,这个alphafold是预测不出来的;6. 由于alphafold时准时不准,因此它还是不能代替实验,不过这丝毫不妨碍它是个强大的辅助工具。
Souce: 纽普生物 2021-07-29