单个氨基酸的变化有可能会剧烈的影响蛋白的活性和功能,除了通过X-ray实验的方法以外。用软件来预测点突变蛋白的结构,解释其功能的变化/无变化,是最常见的蛋白结构分析的工具。
接下来我们会通过一个实际的点突变例子来介绍操作方法。
范例蛋白名称:BARNASE, 点突变E73W
WT蛋白PDB编号:1b27
序列:
>1b27:A|PDBID|CHAIN|SEQUENCE
AQVINTFDGVADYLQTYHKLPDNYITKSEAQALGWVASKGNLADVAPGKSIGGDIFSNREGKLPGKSGRTWREADINYTSGFRNSDRILYSSDWLIYKTTDHYQTFTKIR
Part 1. 用Pymol预测点突变蛋白:
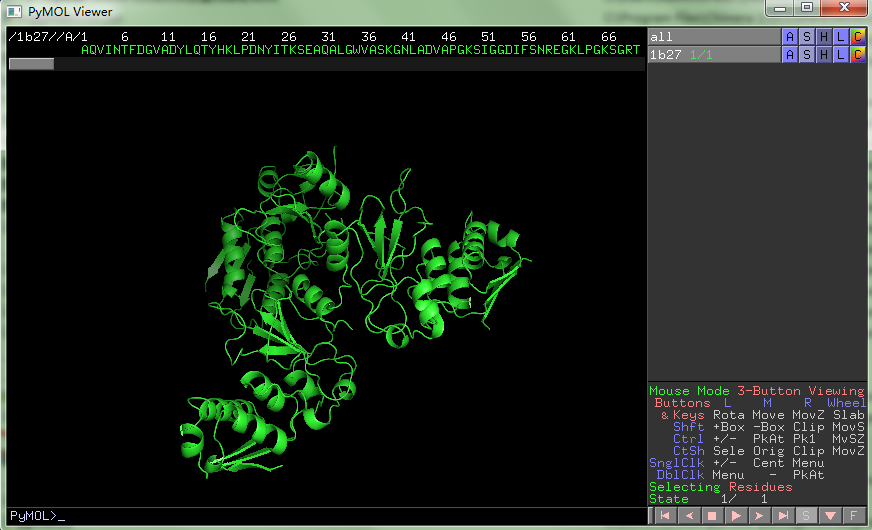
1. Pymol打开1b27.pdb文件,依次操作:Hide -> everything; show -> cartoon; display->sequence, 可以看到:
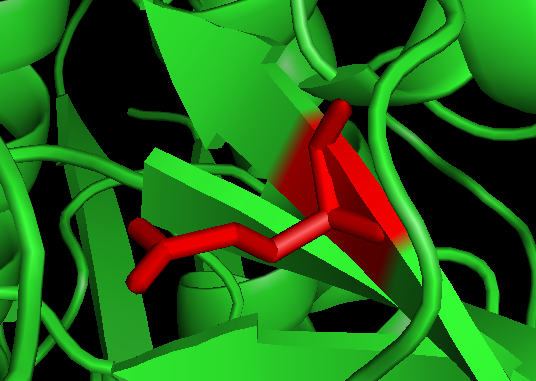
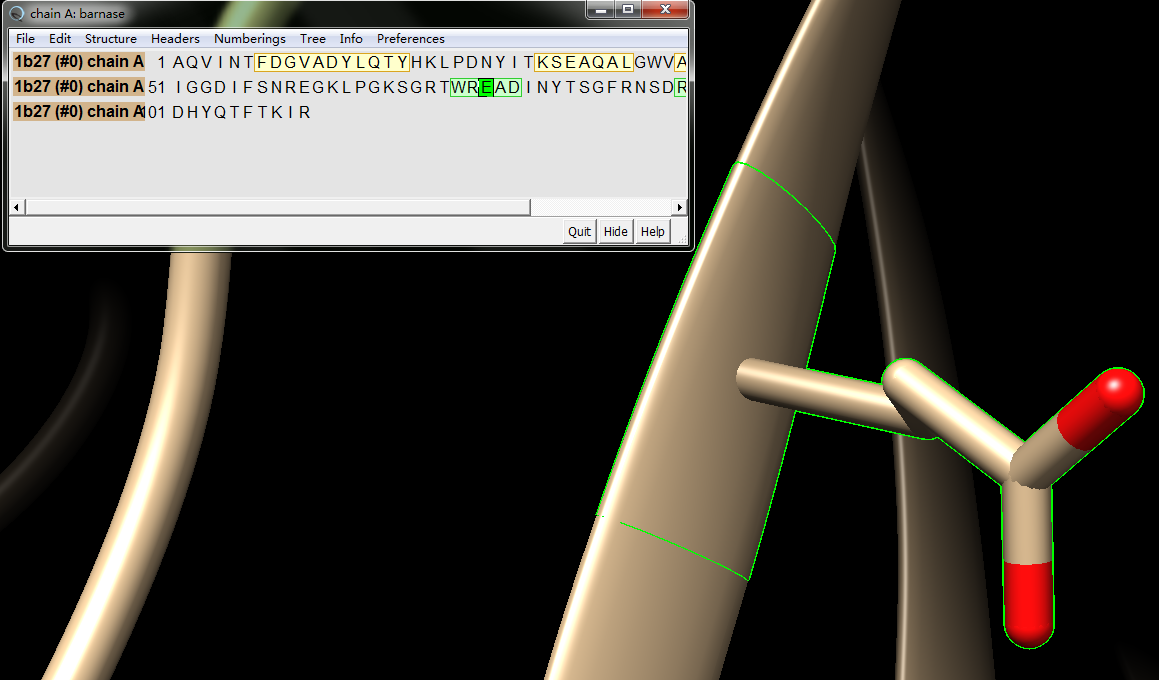
2. 找到Chain A #73, 谷氨酸 Glu E。依次操作,选择氨基酸E; 点show->stick; 点Color->red标红;可以看到该氨基酸E在Pymol被标记成红色而且可以显示侧链:
3. 点Wizard->Mutagenesis; 点 NO-MUTATION, 选择色氨酸 Trp W;Pick-#73 Glu, Color->yellow, 可以看到黄色是点突变以后Trp, 红色是原来的Glu. 点击Apply保存就只剩下黄色的Trp了。

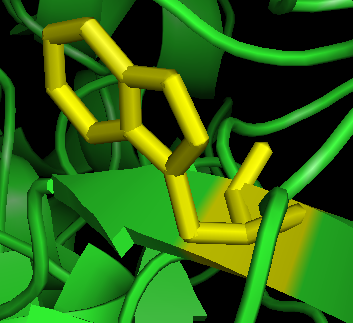
可以看到,做完点突变后,周围残基并没有发生构象上的变化,意味着pymol不会自动做能量最小化处理。
Part 2. 用Chimera做结构点突变:
Chimera相比pymol功能更强大,做点突变可以根据氨基酸不同方向的概率(实验数据)来选择不同的点突变氨基酸结构状态。并且后期通过动力学模拟,来调整点突变以后其他氨基酸结构的变化。
1. file->fetch by ID->输入1b27, fetch蛋白结构;
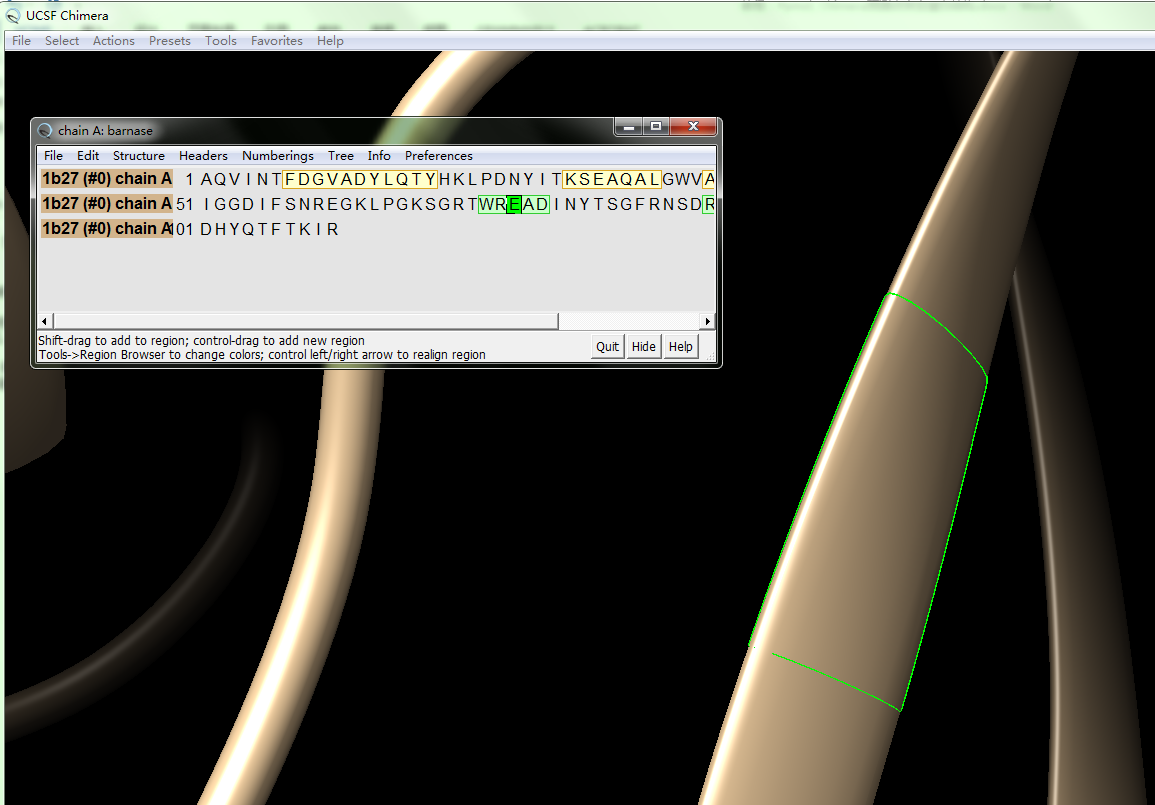
然后点tool->sequence->sequence->选择chain A->show,可以看到Chain A的序列,鼠标左键按住Glu E,右键可以看到:
2. 然后action->atom/bonds->show, 把氨基酸展示出来
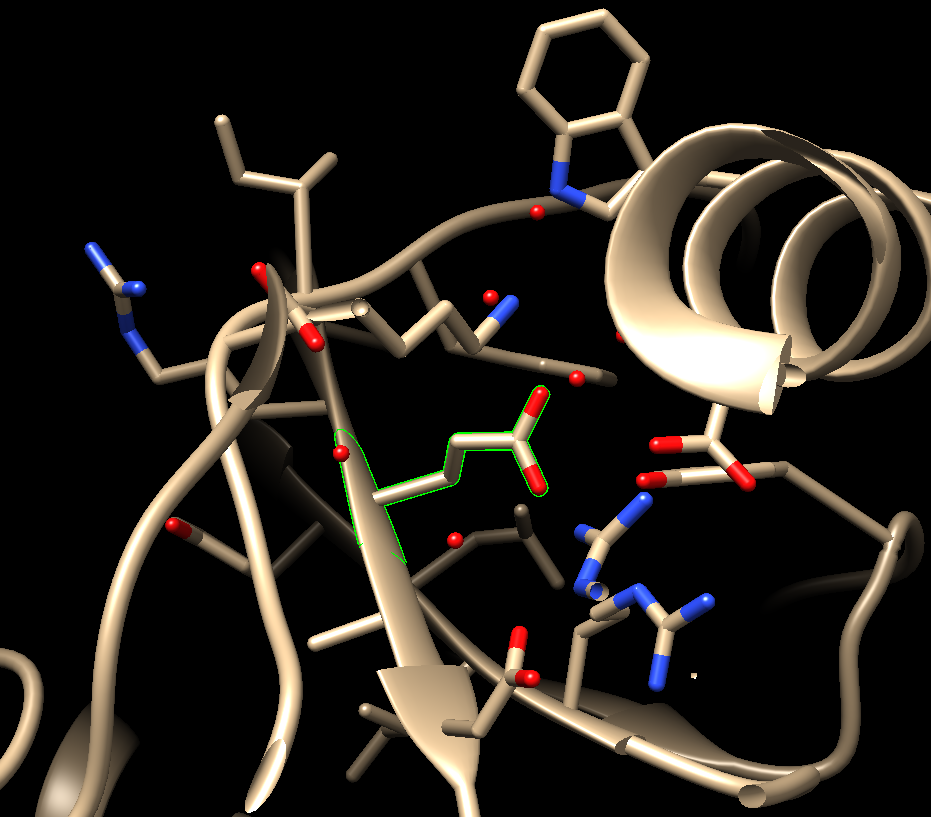
Select->zone->5A范围->打钩->OK, 把目标氨基酸周围的小分子和氨基酸也展示出来,然后还是选择Glu E,可以看到:
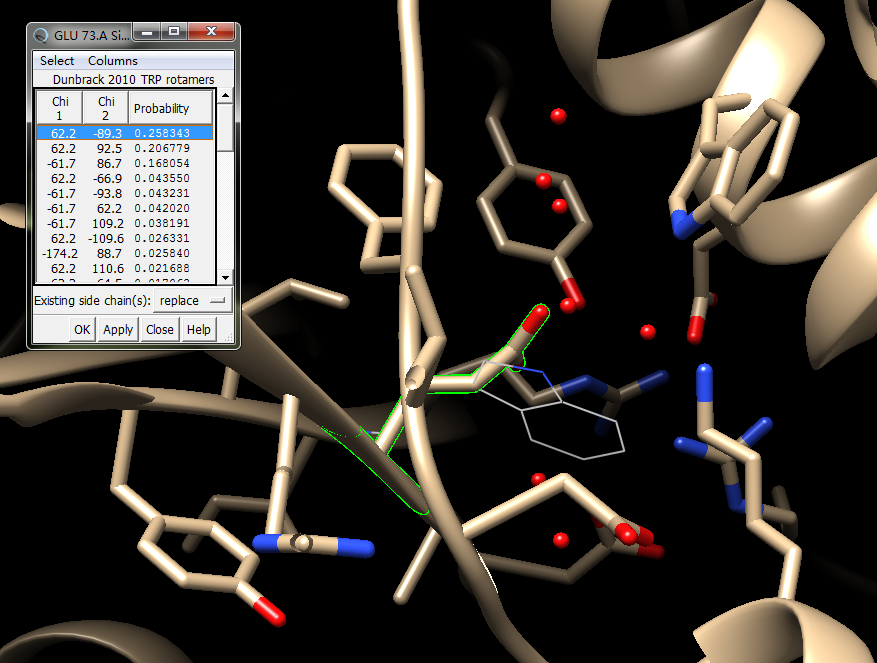
3. 接下来做点突变到TRP:Tools->Structure editing->Rotamers->选择TRP ->Apply
这时候会出来很多种可能的结构,然后有实验统计的概率,选择一个不会碰撞到其他氨基酸的,可能性最大的结构,第一个看起来不错,就是它了:
点击APPLY:
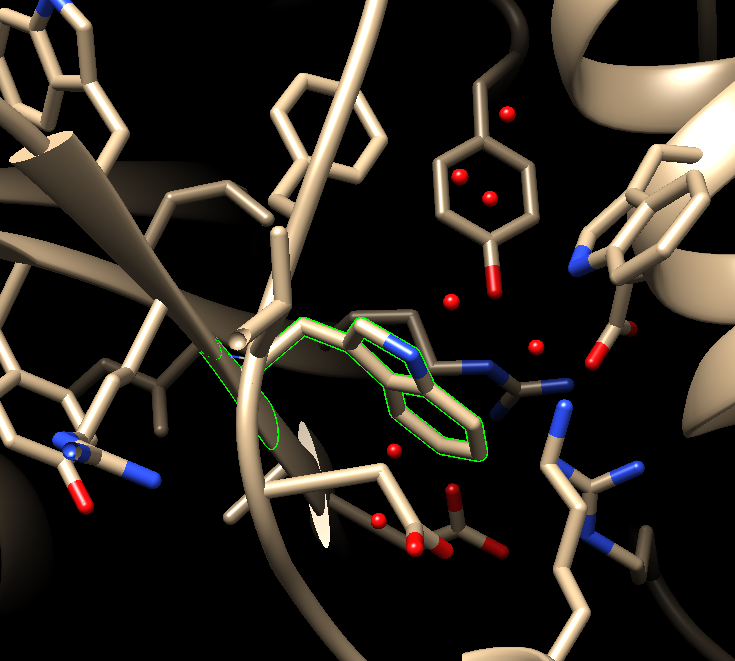
可以很明显的看到周围残基的取向稍有变化,也就是突变后chimera会对模型自动进行能量最小化处理。
另外,对于非标准氨基酸的突变,需要用到一个插件:SwissSidechain Chimera plugin,插件地址:https://swisssidechain.ch/visualization/chimera.php
最后,对于上述操作,其实只用一行命令就可以搞定:swapaa Trp #0:73
表示第一个模型(#0)的73位氨基酸(:73)替换成Trp
Souce: 纽普生物 2019-08-09