DNA甲基化是表观遗传学(Epigenetics)的重要组成部分,在维持正常细胞功能、遗传印记、胚胎发育以及人类肿瘤发生中起着重要作用,是近年来新的研究热点之一。这个可以从国自然中标的结果看出。加上近来对甲基化在肿瘤中重要性的报道,想必这一热潮还有待持续。你是不是也对它垂涎已久,却苦于无从下手,其实甲基化的研究其实并没那么难,一句话,还是套路。
随着高通量测序技术(NGS)技术的发展,使我们能够从全基因组水平来分析5’甲基胞嘧啶及组蛋白修饰等事件,由此能够发现很多传统的基因组学研究所不能发现的东西,这就是所谓的“DNA甲基化测序”!
DNA甲基化测序方法按原理可以分成三大类:
1、重亚硫酸盐测序;
2、基于限制性内切酶的测序;
3、靶向富集甲基化位点测序;
DNA甲基化测序常用方法
基于以上原理又有数种不同的测序方法,下面,就介绍10种DNA甲基化测序的常用方法及参考文献:
1) 重亚硫酸盐测序
该方法可以从单个碱基水平分析基因组中甲基化的胞嘧啶。首先,利用重烟硫酸盐对基因组DNA进行处理,将未发生甲基化的胞嘧啶脱氨基变成尿嘧啶。而发生了甲基化的胞嘧啶未发生脱氨基,因而,可以基于此将经重亚硫酸盐处理的和未处理的测序样本进行比较来发现甲基化的位点。
【相关文献】
Shotgun bisulphite sequencing of theArabidopsis genome reveals DNA methylation patterning.
Highly integrated single-base resolutionmaps of the epigenome in Arabidopsis
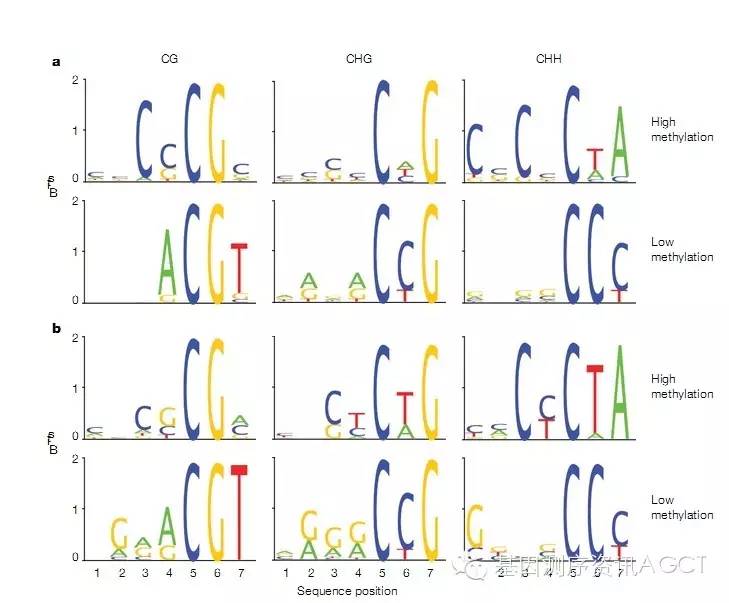
2)重亚硫酸盐处理后接头标记技术(PBAT)
为了避免重亚硫酸盐处理时模板的丢失,通常会在重亚硫酸盐处理后进行接头连接和随机引物的扩增。
【相关文献】
Amplification-free whole-genome bisulfitesequencing by post-bisulfite adaptor tagging
http://www.ncbi.nlm.nih.gov/pubmed/16224102
3)限制性内切酶-重亚硫酸盐靶向测序(RRBS)
该技术是指对基因组上CpG岛或CpG甲基化较密集的区域进行靶向测序。样本首先经几种限制酶进行消化处理,然后经重亚硫酸盐处理,最后再测序。这种方法可以发现单个核苷酸水平的甲基化。
【相关文献】
Reduced representation bisulfite sequencing for comparative high-resolution DNA methylation analysis.
http://nar.oxfordjournals.org/content/33/18/5868.long
4)氧化-重亚硫酸盐测序(oxBS-Seq)
5’羟甲基胞嘧啶(5’hmC)是5’甲基胞嘧啶脱甲基成胞嘧啶过程的中间产物,重亚硫酸盐测序无法对二者进行区分。通过氧化-重亚硫酸盐测序,5’甲基胞嘧啶保留,而5’羟甲基胞嘧啶(5’hmC)被氧化,进而脱氨基成尿嘧啶。通过将经过氧化处理和未处理的样本进行测序比较,即可从单个碱基水平分辨5’羟甲基胞嘧啶(5’hmC)和5’甲基胞嘧啶。
【相关文献】
Quantitative sequencing of 5-formylcytosinein DNA at single-base resolution.
http://www.ncbi.nlm.nih.gov/pmc/articles/PMC4188980/pdf/emss-60510.pdf
5)TET辅助的重亚硫酸盐测序(TAB-seq)
TAB-seq采用葡萄糖亚胺与5’羟甲基胞嘧啶(5’hmC)作用来保护免受TET蛋白的氧化。5’甲基胞嘧啶和未甲基化的胞嘧啶被脱氨基成尿嘧啶,进而可以从单个碱基水平鉴定5’羟甲基胞嘧啶(5’hmC)。
【相关文献】
Base-resolution analysis of 5-hydroxymethylcytosine in the Mammalian genome
6)甲基化敏感性的限制酶测序(MRE-Seq)
MRE-Seq将甲基化作用的敏感性和限制酶的特异性结合起来进而鉴定CpG岛的甲基化状态。
【相关文献】
Genome-scale DNA methylation analysis
7)HELP-Seq
HELP-Seq采用HpaII及其甲基化不敏感的限制性内切酶MSPI处理,来对基因组内及基因组间的甲基化位点进行比较,进而实现甲基化测序。
【相关文献】
Comparative isoschizomer profiling ofcytosine methylation: the HELP assay
8)甲基化DNA免疫共沉淀测序(MeDIP)
MeDIP是一种采用抗体或甲基化DNA结合蛋白来捕获富集甲基化DNA的技术,这种技术可以发现基因组中高度甲基化的区域,如CpG岛,但不能进行单个碱基水平的分析。
【相关文献】
Chromosome-wide and promoter-specificanalyses identify sites of differential DNA methylation in normal andtransformed human cells
9)甲基化结合域捕获技术(MBD-CAP)
MBD-CAP技术利用甲基化DNA能够结合蛋白MeCP2,MBD1-2 和 MBD3LI来对甲基化的DNA进行免疫沉淀。与MeDIP技术相似,该技术也是可以发现基因组中高度甲基化的区域,不能从单个碱基水平分析甲基化。
【相关文献】
High-resolution mapping of DNAhypermethylation and hypomethylation in lung cancer
10)基于探针的靶向富集技术
甲基化测序靶向富集技术采用合成寡核苷酸探针来捕获CpG岛、基因启动子区域以及其他一些显著性甲基化的区域。目前,Agilent 和 Roche Nimblegen公司已有这种商品化的试剂盒。
最后,Pacific Biosciences(Pacbio)公司的这项SMRT DNA测序技术采用动力学原理来直接检测甲基化的胞嘧啶。
甲基化研究方法——甲基化PCR
接下来,就用一个简单的方法说明甲基化研究究竟多么不在话下!也许有的小伙伴已经猜到了,今天要讲的就是甲基化PCR!这种方法灵敏度高,无需特殊仪器,关键是经济实用,是目前应用最为广泛的检测方法。
然而小伙伴们要注意的事,甲基化PCR是环环相扣的,你从预测甲基化位点设计引物到最后的每一步都要小心!甚至在最后PCR的时候,点样的顺序都是和WESTERN BLOT一样有讲究的!
第一步 在线引物设计:http://www.urogene.org/methprimer/
把你基因的启动子序列拷贝至此
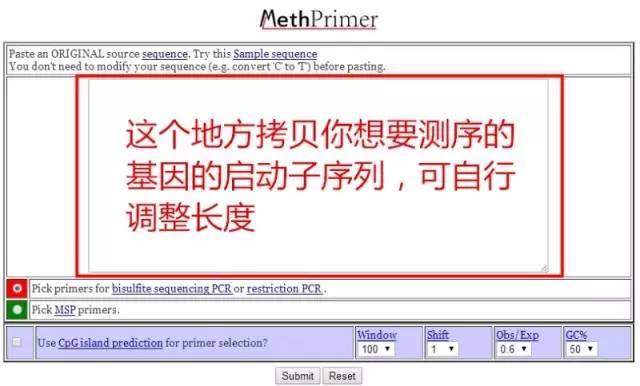
坐等结果,它会预测甲基化位点并设计好甲基化引物(很多对引物供你选择,当然你还要根据设计出来的引物的退火温度是否合理之类的进行筛减),如此智能,拿做己用最为省心了!
第二步 基因组DNA的提取。
这一步完全可以购买供细胞或组织使用的DNA提取试剂盒,大神们自己配试剂提取完全可以。此步重点在于DNA的纯度,即减少或避免RNA、蛋白的污染很重要。因此在提取过程中需使用蛋白酶K及RNA酶以去除两者。
使用两者的注意事项:
1:蛋白酶K可以使用灭菌双蒸水配制成20mg/ml;
2:RNA酶必须要配制成不含DNA酶的RNA酶,即在购买市售RNA酶后进行再处理,配制成10mg/ml。否则可能的后果是不仅没有RNA,连DNA也被消化了。两者均于-20度保存。
第三步 亚硫酸氢钠修饰基因组DNA(也可直接购买试剂盒)
原理:亚硫酸氢钠处理后,DNA中未发生甲基化的胞嘧啶脱氨基转变成尿嘧啶,而甲基化的胞嘧啶保持不变,通过设计出来的特异性甲基化引物和非甲基化引物对PCR取得条带的亮度比较来判断序列是否被甲基化。
大神们请继续自配试剂使用~~(后末附有自配试剂与使用步骤,需要请自行查看)
最后一步 PCR 就不再赘述啦~~
最终如果与预期相符的话,会有如例图所示的结果,M代表甲基化引物,U代表非甲基化引物,1和2分别是肺癌组织和正常组织所提取的基因组。可见在肺癌组织中甲基化程度明显偏高。

图片引自:Xu Zhai, Shi-Jun Li. Methylation of RASSF1A and CDH13 Genes in Individualized Chemotherapy for Patients with Non-small CellLung Cancer. Asian Pac J Cancer Prev, 15 (12), 4925-4928
这样的检测并不局限于组织,经过药品(如甲基化抑制剂)处理后的肿瘤细胞与未处理的细胞比较某基因甲基化程度也是可行的。另外,小伙伴们如果手头样本量够大,可以不局限于比较癌组织和癌旁组织的差异,不妨对癌组织样本分期,或者对病人分类(如初发肿瘤和复发肿瘤病人)来检测有无甲基化差异,也许结果会有惊喜!
附:自配试剂的使用步骤及注意事项
1:将约2 ug DNA于1.5 ml EP管中使用DDW稀释至50 ul;
2:加5.5 ul新鲜配制的3 M NaOH;
3: 42℃水浴30 min;
水浴期间配制:
4:10 mM对苯二酚(氢醌),加30 ul至上述水浴后混合液中;(溶液变成淡黄色)
5: 3.6 M亚硫酸氢钠(Sigma,S9000),配制方法:1.88 g亚硫酸氢钠使用DDW稀释,并以3 M NaOH滴定溶液至PH 5.0,最终体积为5 ml。这么大浓度的亚硫酸氢钠很难溶,但加入NaOH后会慢慢溶解,需要有耐心。PH一定要准确为5.0。加520 ul至上述水浴后溶液中。
6:EP管外裹以铝箔纸,避光,轻柔颠倒混匀溶液。
7:加200 ul 石蜡油,防止水分蒸发,限制氧化。
8:50℃避光水浴16 h。
(时间安排小tip:此步可以在4 pm开始做,熟练的话不到5 pm即可完成,水浴16 h正好至次日8 am以后收,这样不用熬夜时间的安排是多么让人心旷神怡。)
注意事项:
1:基因组DNA的量不需十分精确,宁多勿少,因为在以后纯化回收步骤中会有丢失,且此方法修饰最多可至4 ug。
2:所有试剂均须新鲜配制,所以配液的技术要过关,既要快,又要精确。
3:亚硫酸氢钠溶液呈强酸性,一定用碱将PH调制5.0,否则PH不合适会影响后续纯化吸收。
4:水浴最好达16小时,虽可以短至8小时,但后者修饰会有不完全。
Souce: 解螺旋 2016-07-08