qPCR引物与普通PCR引物的设计虽然在设计原则和方法上大致相同,但还是有些不一样的地方,严格的qPCR引物设计一般要求其中一条引物要夸外显子,最好在3‘端设计引物,产物的长度在300bp以内等。小编今天给大家介绍的这个在线软件Primer3 Plus和Oligo Calc,可以满足大家对qPCR引物设计日益增高的要求。
第一步:从数据库Ensembl:http://asia.ensembl.org/index.html中获得基因的cDNA序列。
1. 本文以ApoE基因为例,输入目的基因
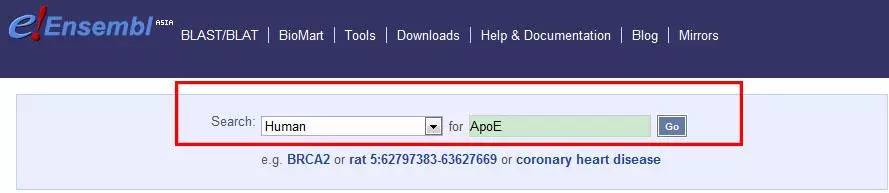
2. 在左侧栏选择Transcript
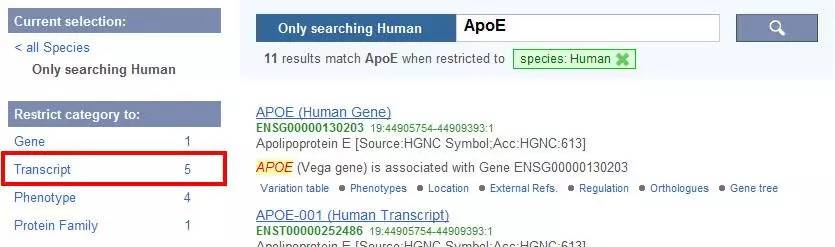
3. 选择APOE-001中的Transcript进入,点击左侧cDNA
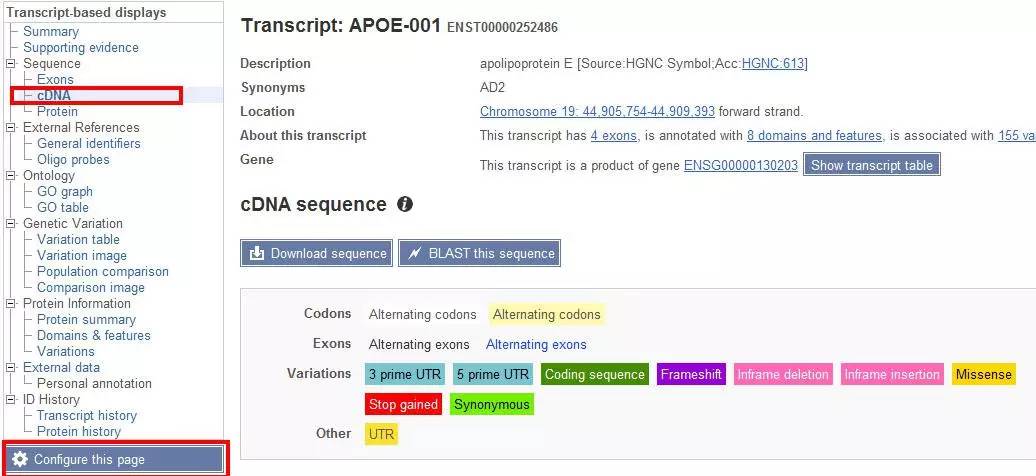
4. 然后点击CONFIGURE THIS PAGE进入,设置你要显示的内容, 在SHOW EXONS这一项打个勾,然后点击“Save configuration as”保存序列
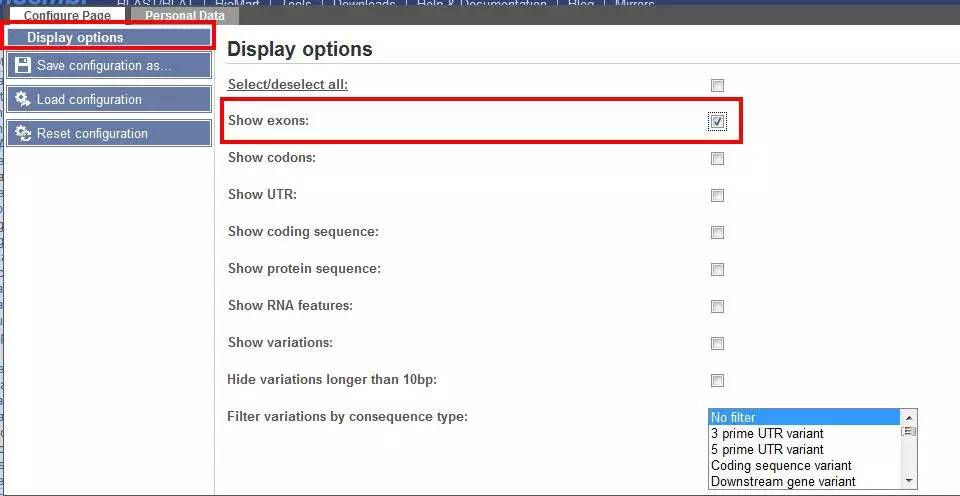
5. 以RTF的格式DOWNLOAD下来,可以Word的方式打开,不同的颜色代表不同外显子的间断

第二步:利用Primer3 Plus获取引物,打开Primer3 Plus在线软件:http://www.primer3plus.com/cgi-bin/dev/primer3plus.cg
1. 打开Primer3 Plus,将序列粘贴在指定位置
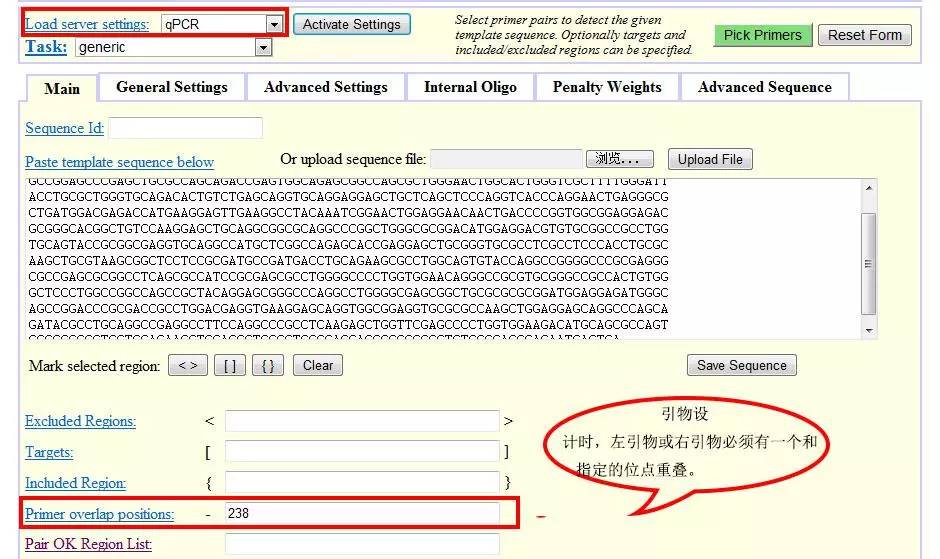
2. 在Load server settings项目中选择“qPCR”:
因为设计为避免因基因组DNA的污染而P出以基因组DNA为模板的产物,故设计引物时其中一个引物最好能够夸外显子设计,故在Primer overlap position中输入左引物或右引物必须重叠的位点,本文在序列238位氨基酸是后两个外显子的连接处,故输入238。或者可以在Pair OK Region List中,作相应的设置,该标签的值为多个,值和值之间用“;“隔开,单个值为:<left_start>,<left_length>,<right_start>,<right_length>。比如:SEQUENCE_PRIMER_PAIR_OK_REGION_LIST=100,50,300,50 ;900,60,, ; ,,930,100
表明有引物设计有三种选择:
左引物在100~150bp区间进行设计,右引物在300~350bp的区间进行设计;
左引物在900~960bp区间进行设计,右引物随意;
右引物在930~1030bp区间进行设计,左引物随意。
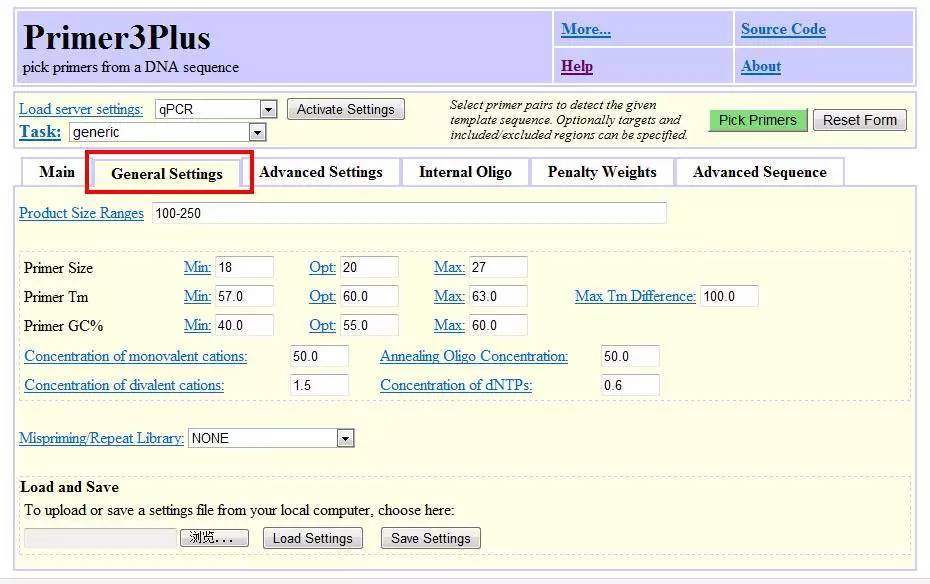
3. 然后在General Settings设置引物长度、引物Tm值、GC含量等参数。
4. 如果对引物有更多的设计要求:
可以在Advanced Settings、Internal Oligo、Penalty、Andvanced Sequence中设置相关的参数。如无则可点击绿色按钮“Pick Primers”,获得引物。
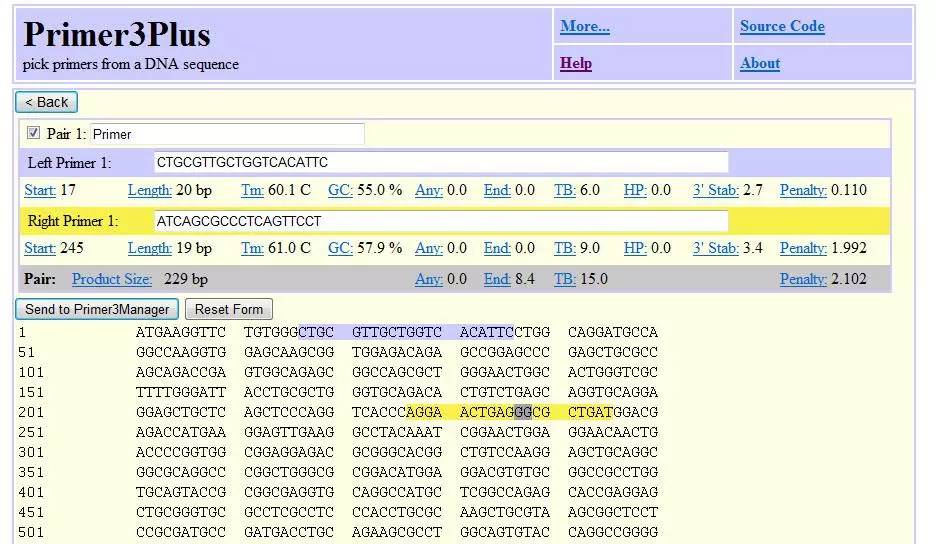
第三步:分析引物
1. 打开Oligo Calc网页,将正向引物粘贴到指定位置

2. 点击Check Self-complmentarity,检测引物自身是否形成互补二聚体、发夹结构等。

3. 结果如下图所示:

4. 重复步骤1至3,粘贴反向引物。
5. 点击mfold
将包含正反引物之间的序列即产物放到mfold,查看产物是否折叠形成二级结构。如结果显示产生二级结构,则可重新在Primer3 plus改变条件重新获得qPCR引物。
6. 最后点击BLAST,检测引物的特异性。
经过层层筛选,最后得到符合条件的引物实在是凤毛麟角,所谓鱼和熊掌不可兼得,所以可能得将就一下接受各方面都不错但还是有一点不完美的引物,而且引物好不好用,也只有realtime PCR以后才能得到验证。所以,请大胆地去pick primers吧!
作者:解螺旋.翠花解螺旋出品
2015-07-22