作者:解螺旋.子非鱼
RNA自从在中心法则中占有一席之位以后,大家就开始争相热捧。RNA的单链性质导致它与DNA不同,分子中的一部分核苷酸可与其他部分的核苷酸互相配对,即RNA可产生折叠。我们知道生物分子的功能与结构是密切相关的,折叠的RNA能部分甚至完全解释RNA分子的功能。对于当前的三维结构还是过于复杂,所以我们主要专注于二级结构的预测。
有关二级结构的预测,小鱼今天就为大家介绍一款性价比极高的软件:RNAStructure。
RNAStructure利用 Zuker算法(Zuker Algorithm),根据最小自由能原理 (minimizing free energy), 通过RNA一级序列预测RNA二级结构。 预测所用的热力学数据是最近由Turner实验室获得。该软件使用了一些模块来扩展Zuker算法的能力,并使之为一个界面友好的 RNA折叠程序。
为什么说他性价比高呢?除了可以检测RNA单链折叠,还可以进行DNA单链折叠、RNA或DNA分子间折叠、还有高大上的Refold 功能:重新折叠选项可读取一个由任何折叠模块生成的存盘文件*.sav,并生成一个不同的次优结构。(就是“折上再折”,折到你满意为止!)
小鱼今天就给大家介绍如何使用RNA structure对RNA进行二级结构的预测。
打开RNAstructure,点File-new sequence,将序列粘贴到指定的位置,命名并保存序列。
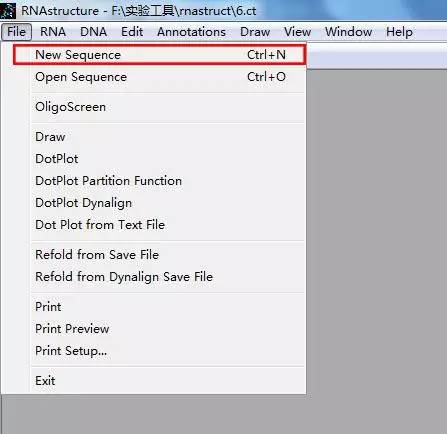
点击RNA一栏,可以看到许多重要的菜单,比如:
Fold RNA Single Strand:此项使用 RNA 参数打开折叠窗口;
Fold RNA Biomolecular:此项使用 RNA 参数折叠两个单体;
MaxExpect: Predict RNA EMA Structure:此项可预测最大期望精确度的RNA结构;
Efn2 RNA:此项测定生成的 RNA 二级结构的自由能,储存在 ct 文件格式中;
Dot Plot:此项可计算任何折叠的序列的点阵图,用于已经折叠并存盘的序列;
Oligo Walk:打开 Oligo Walk tool 帮助确定一个与 RNA 对象紧密杂交的寡聚体;
MaxExpect:Predict RNA EMA Structure;
本例选择进行RNA单链折叠,点击“Fold RNA Single Strand”:
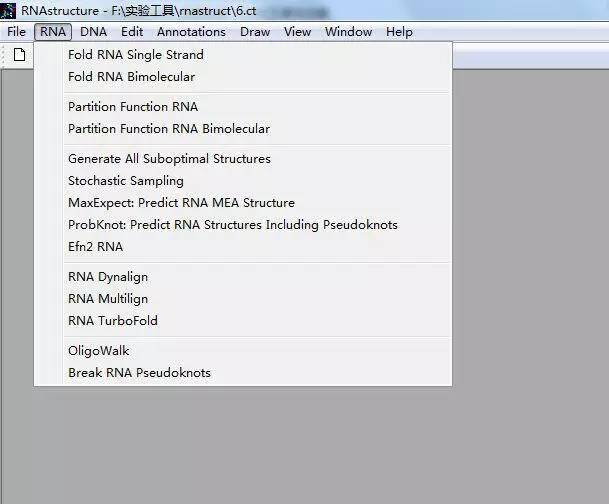
弹出RNA Fold窗口,点击“Sequence File”选择要预测的序列,窗口还有以下重要选项:
Max% Energy Difference(最大能量差异百分值):设定所允许的输出结构自由能与最小自由能的差异百分数数值。例如:如果结构的最小自由能为-100 kcal/mol,最大能量差异百分值,则会排除任何等于或大于-90 kcal/mol 的结构(大于即为负值更小)。
Max number of structures(最大结构数):所预测的可能结构的数量的绝对上限值。最大为1000。
Window size(窗口大小):此参数设定所生成的次优结构之间的差异程度。Windows size设定值越小,则生成的结构则越接近的结构;反之,Windows size设定值越高,则生成的结构则差异越大。
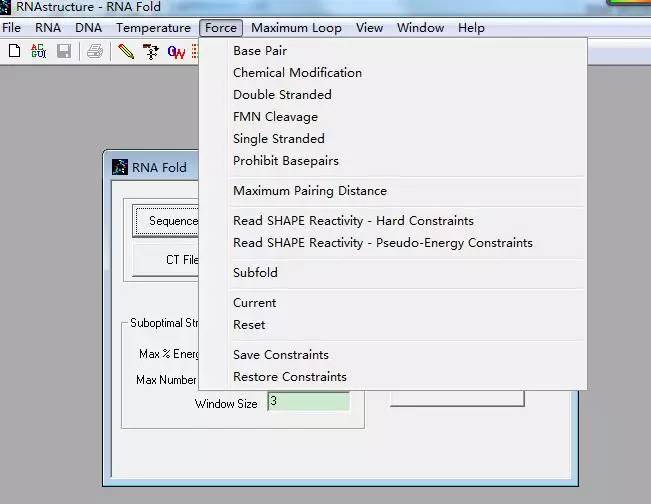
在菜单栏点击Force:强制选项,就是各种高级设置,只为打造更个性化更special的RNA结构!比如:
Base pair:强制某个碱基配对;
Chemical Modification:强制某个碱基进行化学修饰;
Double Stranded:强制某个碱基为双链模式
不过筛选条件越苛刻,获得合适的RNA二级结构就越少甚至没有
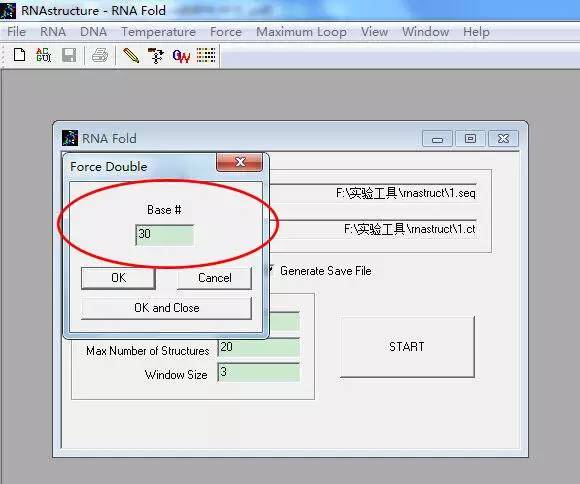
此处选择强制第30个碱基为双链模式,点击Double Stranded,弹出“Force double”窗口,在指定位置输入“30”,点击“OK and Close”:
点击“Temperature”和“Maximum Loop”可以设置温度和最大茎环数~
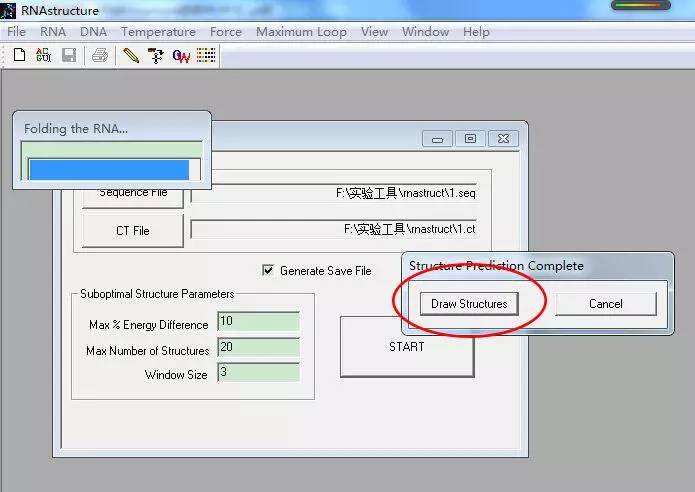
经历了这么多的繁文缛节,终于到高潮了!点击RNA Fold窗口中的“START”,再点击“Draw Structures”:
千呼万唤,终于粉墨登场,我的RNA二级结构:
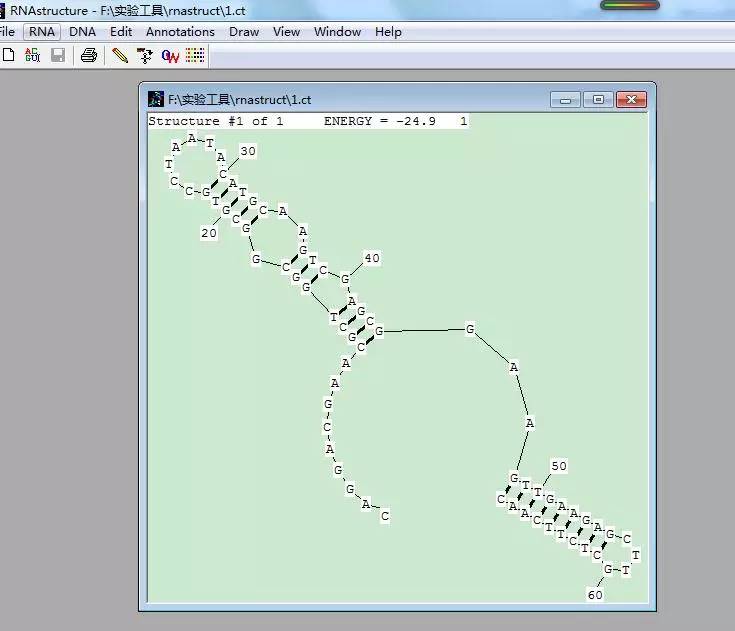
最后,选择Edit-Copy 菜单将结果图拷贝到剪贴板,并可复制到其它应用程序。
RNA二级结构的预测,听起来好高大上,跟着小鱼操作一遍,你会发现其实很简单哦!
2015-07-22